 Palline e molle: a volte una soluzione semplice può eguagliare tecniche molto complicate di analisi delle molecole. Un gruppo di scienziati della SISSA ha proposto un’alternativa rapida e basata su principi molto semplici per predire la dinamica interna delle molecole di RNA (in che modo le diverse parti si muovono le une rispetto alle altre). Il modello offre risultati comparabili a quello di altre tecniche già in uso ma molto più complesse e dispendiose dal punto di vista computazionale. La ricerca è stata pubblicata sulla rivista Nucleic Acids Research.
Palline e molle: a volte una soluzione semplice può eguagliare tecniche molto complicate di analisi delle molecole. Un gruppo di scienziati della SISSA ha proposto un’alternativa rapida e basata su principi molto semplici per predire la dinamica interna delle molecole di RNA (in che modo le diverse parti si muovono le une rispetto alle altre). Il modello offre risultati comparabili a quello di altre tecniche già in uso ma molto più complesse e dispendiose dal punto di vista computazionale. La ricerca è stata pubblicata sulla rivista Nucleic Acids Research.
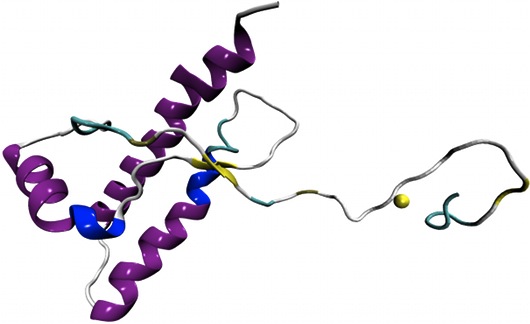
Si chiama Elastic Network Model (ENM), e permette di predire, praticamente in tempo reale (i tempi per il processamento sono di pochi secondi), la dinamica interna delle molecole di RNA. Per comprendere il funzionamento dell’RNA, è infatti utile conoscere la composizione della sequenza di nucleotidi, la forma tridimensionale della molecola, ma anche la sua dinamica interna, e cioè come le diverse parti della molecola si muovono fra loro: quali sono rigide e quali elastiche, quali sono connesse e quali indipendenti, e via dicendo. Per conoscere questo aspetto si usano tecniche come la “Dinamica Molecolare”, un metodo di simulazione al computer che al momento è giudicato come il più accurato, che però ha l’inconveniente di essere molto dispendioso dal punto di vista computazionale. A volte un calcolo può richiedere giorni o settimane.
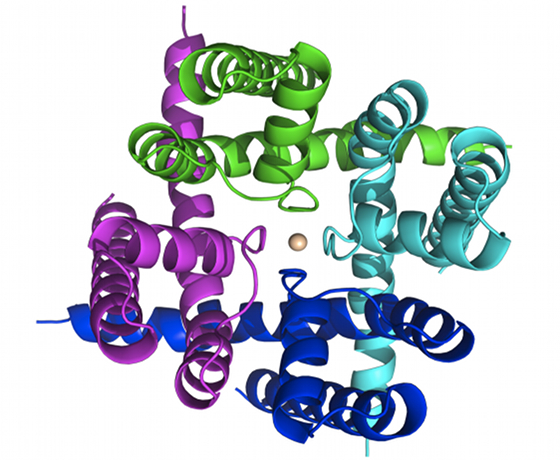
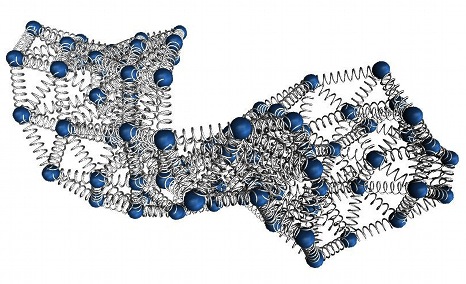
Il gruppo coordinato da Giovanni Bussi, della Scuola Internazionale Superiore di Studi Avanzati (SISSA) di Trieste, in collaborazione con Cristian Micheletti, anche della SISSA, ha costruito un ENM, un modello alternativo, basato su regole semplici. Nel modello della SISSA gruppi di decine di atomi sono rappresentati come delle “palline” che poi sono collegate da molle che mantengono l’intelaiatura strutturale della molecola.
“A dispetto della sua semplicità, il nostro modello è in grado di predire le fluttuazioni strutturali di molecole di RNA con altrettanta accuratezza di modelli più complicati” spiega Bussi. Nei test l’ENM ha dato risultati quasi indistinguibili dalla Dinamica Molecolare, e dunque potrebbe rappresentare una valida alternativa a questa metodologia. Oltre a Bussi e Micheletti hanno partecipato alla ricerca anche Giovanni Pinamonti e Sandro Bottaro, entrambi della SISSA.
Più in dettaglio…
Oltre alla parte teorica del lavoro il team ha anche eseguito un confronto con dati sperimentali. Gli esperimenti di reattività SHAPE usano dei reagenti che “attaccano” i nucleotidi più mobili nella molecola di RNA. Sequenziando la molecola prima e dopo la reazione si identificano i nucleotidi che hanno reagito, ossia quelli che nella molecola erano più liberi di muoversi. Questa tecnica sperimentale permette dunque di avere informazioni sulla dinamica della molecola. Bussi e colleghi hanno confrontato le previsioni dell’Elastic Network Model con i dati sperimentali, trovando anche qui un sostanziale accordo.